クラスター分析は
- 距離行列の生成(類似度行列ではない!)
- クラスタリングの実行
という流れになる。
それぞれのステップで、採用する
- 距離の種類
- クラスタリングの方法
がチューニング変数となる。
この順に手順を見ていく。
行数、列数の多いビッグデータ向きのデータ形式であるMatrixパッケージに対応した距離行列についても説明する。
目次
距離行列を生成する
類似度行列ではなく距離行列を作る。similarityではなくdistanceを作る。
直感的にはデータから距離の指標(どれだけ離れているか)ではなく類似度(どれだけ近いか)の指標を抽出し、そこからクラスタリングしたいケースが多いのだが、あくまで類似度指標に基づいた距離行列を生成するのである。
通常のdist関数では対応する距離の種類が少ないのでproxyパッケージを使う。
距離行列を生成するメソッドはdist(data, method="距離の種類")。
require(proxy)
dcos <- proxy::dist(USArrests, method="cosine")
戻り値はdistオブジェクト。行列やデータフレームとは異なるので扱いに注意。
distオブジェクトは同じ次元なら加減乗除が可能。たとえばdist1, dist2ともにdistオブジェクトの場合、
dist1 + dist2
dist1 - dist2
dist1 * dist2
dist1 / dist2
いずれも対応する要素を加減乗除したdistオブジェクトとして返ってくる。
類似度の種類
類似度(どれだけ近いか)の種類と、それに対応したdist()関数で指定する引数methodの値
- 数値ベクトルに対するもの
- コサイン類似度:
cosine - 拡張Jaccard:
eJaccard - fuzzy Jaccard:
fJaccard - Pearsonの積率相関係数:
correlation
- コサイン類似度:
- 0/1値に対するもの(集合に入る or not)
- Jaccard係数:
Jaccard - Simpson係数:
Simpson - Dice係数:
Dice - Simpson-Jaccard係数は直接算出できないが、
(dSimpson + dJaccard) / 2とするだけでSimpson-Jaccardの距離行列を取得できる
- Jaccard係数:
- 2×2の分割表に対するもの
- ファイ係数:
Phi - ユールのQ:
Yule
- ファイ係数:
(参考)主な類似度指標の違い
数値ベクトルの類似度では、
Pearsonの積率相関係数は、各成分から平均値を引いたもののcos類似度と一致する
2値変数の類似度指標は基本的に
共通部分2集合の【何か】
【何か】が指標によって異なる。
- Jaccard係数→和集合
- Simpson係数→共通部分
- Dice係数は→算術平均
Simpson-Jaccard指数はSimpson指数とJaccard指数の算術平均。
Diceは分母が算術平均になるという違いがある(調和平均に似ている)。
距離の種類→数値ベクトルに対するもの
距離(どれだけ離れているか)の種類と、それに対応したdist()関数で指定する引数methodの値
- ユークリッド距離:
Euclidean - マハラノビス距離:
Mahalanobis - マンハッタン距離:
Manhattan - チェビシェフ距離:
Chebyshev - ミンコフスキー距離:
Minkowski - キャンベラ距離:
Canberra
たとえば
# コサイン類似度に基づく距離
dcos <- proxy::dist(USArrests, method="cosine")
# 連続量版Jaccard係数
djcd <- proxy::dist(USArrests, method="eJaccard")
# ピアソンの積率相関係数に基づく距離
dcor <- proxy::dist(USArrests, method="correlation")
などとなる。
Matrix形式の行列に対する類似度の算出関数
Rの通常の行列やデータフレームではディメンションの大きいsparse matrix(疎行列)を扱うのが困難である。
Matrixというパッケージは行列を独自の形式で扱うパッケージで、主にsparse matrixなどを扱うのに強い。
このMatrix形式の行列に対して類似度を計算する関数を作った。
jcdSparse(): バイナリデータに対するJaccard係数smpSparse(): バイナリデータに対するSimpson係数ejcdSparse(): 連続量に拡張したJaccard係数esmpSparse(): 連続量に拡張したSimpson係数cosSparse(): コサイン類似度
jcdSparse <- function(X){
X <- as(X, "dgCMatrix")
X@x <- rep(1, length(X@x)) # 非ゼロ要素はすべて1に置換
vec_sum_col <- colSums(X)
AB <- crossprod(X)
sim <- AB / (vec_sum_col - t(AB - vec_sum_col)) # まとめて
return(sim)
}
smpSparse <- function(X){
X <- as(X, "dgCMatrix")
X@x <- rep(1, length(X@x)) # 非ゼロ要素はすべて1に置換
vec_sum_col <- colSums(X)
AB <- crossprod(X)
n <- length(vec_sum_col)
A <- matrix(rep(vec_sum_col, n), n)
B <- t(A)
mat_A_lt_B <- A < B
sim <- AB / (A * mat_A_lt_B + B * (1 - mat_A_lt_B))
return(sim)
}
ejcdSparse <- function(X){
X <- as(X, "dgCMatrix")
vec_xx <- colSums(X^2)
mat_xy <- crossprod(X)
sim <- mat_xy / (vec_xx - t(mat_xy - vec_xx)) # まとめて
return(sim)
}
esmpSparse <- function(X){
X <- as(X, "dgCMatrix")
vec_xx <- colSums(X^2)
mat_xy <- crossprod(X)
n <- length(vec_xx)
mat_xx <- matrix(rep(vec_xx, n), n)
mat_yy <- t(mat_xx)
mat_x_lt_y <- mat_xx < mat_yy
sim <- mat_xy / (mat_xx * mat_x_lt_y + mat_yy * (1 - mat_x_lt_y))
return(sim)
}
cosSparse <- function (X) {
if(class(X)[[1]] != "dgCMatrix") {
X <- as(X, "dgCMatrix")
}
X_normalized_col <- X %*% Diagonal(x = 1 / sqrt(colSums(X^2))) # まとめて
sim <- crossprod(X_normalized_col)
return(sim)
}
距離行列と類似度行列の関係
- 類似度は一致するときに1をとる。独立なら0、正反対なら-1
- 距離は大きいほうが離れている
distance = 1 - similarity
Pearsonの積率相関係数から距離行列を生成するのもこの原理。
(参考)
- 類似度と距離の概念
http://wikiwiki.jp/cattail/?%CE%E0%BB%F7%C5%D9%A4%C8%B5%F7%CE%A5 - その他の距離
http://www.nysol.sakura.ne.jp/mcmd/jp/sect-msim.html - 直接実装するなら
http://sucrose.hatenablog.com/entry/2012/11/30/132803 - 距離に関するRのライブラリと関数
http://www.slideshare.net/yuifu/ss-11776646 - {proxy}パッケージに実装されている距離と類似度の一覧
https://cran.r-project.org/web/packages/proxy/vignettes/overview.pdf
主成分分析とクラスタリング
主成分分析のprcomp()関数についておさらい
prcomp$xが主成分得点行列prcomp$sdevが固有値prcomp$rotationが固有ベクトルbiplot(prcomp)でプロット- 主成分得点×固有ベクトル=相関行列(
scale=Tで実行した場合) prcomp()とprincomp()関数の違い:prcomp()関数:主成分得点を求める際に、主成分得点の不偏分散が固有値に一致するように計算されるprincomp()関数:主成分得点の標本分散が固有値に一致するように計算される
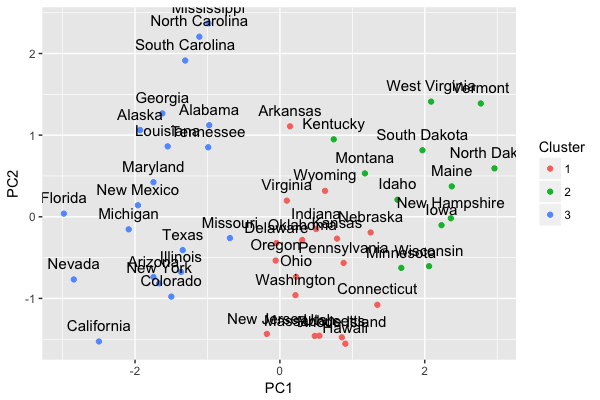
主成分分析を行い、第1主成分と第2主成分をクラスタリング
# 主成分分析
USArrests.prcomp <- prcomp(USArrests, scale = TRUE)
## 第1主成分から第2主成分の主成分得点を抽出
USArrests.scores <- USArrests.prcomp$x[,1:2]
クラスタリング
# 3個のクラスタに分類
USArrests.kmeans <- kmeans(USArrests.scores, centers = 3)
# クラスタサイズ確認
table(USArrests.kmeans$cluster)
プロット
# 第1主成分得点
USArrests.pc1 <- USArrests.prcomp$x[,1]
# 第2主成分得点
USArrests.pc2 <- USArrests.prcomp$x[,2]
# クラスタ
USArrests.clusters <- USArrests.kmeans$cluster
# plot関数でプロットする場合
plot(USArrests.pc1, USArrests.pc2, xlab = "PC1", ylab = "PC2", col = USArrests.clusters)
## ggplot2でプロットする場合
USArrests.pc.df <- data.frame(
State = rownames(USArrests),
PC1 = USArrests.pc1,
PC2 = USArrests.pc2,
Cluster = factor(USArrests.clusters)
)
ggplot(USArrests.pc.df, aes(x = PC1, y = PC2, label = State, color = Cluster)) +
geom_point() + geom_text(size = 4, colour = "black", vjust = -1)
http://www.statmethods.net/advstats/cluster.html

クラスタリングの実行
階層的クラスタリング
ユーザ分類などビッグデータには不向き。
階層的クラスタリングを実行するメソッドはhclust(dist, method="クラスタリングの方法")。
引数で与えるのは距離行列
hclust()の引数methodの種類
- 最近隣法:
single - 最遠隣法:
complete - 群平均法=UPGMA:
average - 重心法=UPGMC:
centroid - メディアン法=WPGMC:
median - Ward法=最小分散法(郡内分散と群間分散の比を最大化):
ward.D2 - McQuitty法=WPGMA:
mcquitty
デンドログラムの生成
距離の種類×クラスタリングの手法で比較するよね。
- コサイン類似度の距離行列
dcos - Jaccard係数の距離行列
djcd - Simpson係数の距離行列
dsmp - Tanimoto係数の距離行列
dtnm - Dice係数の距離行列
ddce
に対して
- Ward法
- 群平均法(UPGMA)
- McQuitty法
で階層クラスタリングした結果をデンドログラムで比較する。
pdf("hclust.pdf")
op <- par(mfrow=c(2,3))
{ # Ward法
wd.dcos <- hclust(dcos, method="ward.D2")
wd.djcd <- hclust(djcd, method="ward.D2")
wd.dsmp <- hclust(dsmp, method="ward.D2")
wd.dtnm <- hclust(dtnm, method="ward.D2")
wd.ddce <- hclust(ddce, method="ward.D2")
plot(wd.dcos, main="cosine similarity/Ward")
plot(wd.djcd, main="Jaccard index/Ward")
plot(wd.dsmp, main="Simpson index/Ward")
plot(wd.dtnm, main="Tanimoto index/Ward")
plot(wd.ddce, main="Dice index/Ward")
}
par(op)
op <- par(mfrow=c(2,3))
{ # 群平均法(UPGMA)
ga.dcos <- hclust(dcos, method="average")
ga.djcd <- hclust(djcd, method="average")
ga.dsmp <- hclust(dsmp, method="average")
ga.dtnm <- hclust(dtnm, method="average")
ga.ddce <- hclust(ddce, method="average")
plot(ga.dcos, main="cosine similarity/UPGMA")
plot(ga.djcd, main="Jaccard index/UPGMA")
plot(ga.dsmp, main="Simpson index/UPGMA")
plot(ga.dtnm, main="Tanimoto index/UPGMA")
plot(ga.ddce, main="Dice index/UPGMA")
}
par(op)
op <- par(mfrow=c(2,3))
{ # McQuitty法
mq.dcos <- hclust(dcos, method="mcquitty")
mq.djcd <- hclust(djcd, method="mcquitty")
mq.dsmp <- hclust(dsmp, method="mcquitty")
mq.dtnm <- hclust(dtnm, method="mcquitty")
mq.ddce <- hclust(ddce, method="mcquitty")
plot(mq.dcos, main="cosine similarity/McQuitty")
plot(mq.djcd, main="Jaccard index/McQuitty")
plot(mq.dsmp, main="Simpson index/McQuitty")
plot(wd.dtnm, main="Tanimoto index/McQuitty")
plot(wd.ddce, main="Dice index/McQuitty")
}
par(op)
dev.off()
分類
# 実際にクラスタ数を指定して、どのIDがどのクラスタの分類されるかベクトルで
cutree(wd.dcos, k=15)
# N行1列の行列に変換してデータフレームにマージ
cbind(df, cluster = matrix(cutree(wd.dcos, k=15), nc=1))
参考
http://rstudio-pubs-static.s3.amazonaws.com/10521_be5bce1e68f44c359bc88e6b0a07ef5c.html
http://www.antecanis.com/texts/group_05/
非階層クラスタリング
k-means
- ハードクラスタリング
- k 個のクラスタのいずれか1個に所属
- Rではの
kmeans(データ, centers = クラスタ数, iter.max = 反復回数){stats}(デフォルトでインストールされているパッケージ)で実行 - 引数で与えるのはデータ
\sum_{l=1}^k \sum_{x_i \in X_l} \parallel x_i – \mu_l \parallel^2 \rightarrow min!
ただし
k : 分割数
x_i : 観測
X_l : クラスタ l
\mu_l : クラスタ l の重心
# デフォルトでは反復回数が10と少ないので指定して実行する
USArrests.km <- kmeans(USArrests, centers = 5, iter.max = 100)
# どのIDがどのクラスタの分類されるかベクトルで
USArrests.km$cluster
# データフレームにマージ
USArrests.km <- cbind(USArrests, cluster = USArrests.km$cluster)
plot.kmeans(kmeans, data) {useful}はkmeansオブジェクトをそのままプロットできるので便利。
require(useful)
plot(USArrests.km, USArrests)
階層型クラスタリングからのk-means
初期値(クラスタの中心点)を階層型クラスタリングで算出し、それに基づいてk-meansをかける方法。
# 階層型クラスタリングからクラスタ数を指定して分類する(コサイン類似度、ウォード法)。
clusters <- cutree(hclust(dist(USArrests, method = 'cosine'), method = 'ward.D2'), k = 5)
# クラスタiの中心点を算出
clust.centroid = function(i, dat, clusters) {
ind = (clusters == i)
colMeans(dat[ind,])
}
# 全クラスタに適用
centers <- sapply(unique(clusters), clust.centroid, USArrests, clusters)
# k-meansの実行。sapply()の結果だと行と列が逆なので、転置して引数に与える
kmeans(USArrests, centers=t(centers))
参考
https://www.biostars.org/p/13143/
k-meansの初期値問題の一つの解になれば。
fuzzy c-means
- ソフトクラスタリング
- 全部で c 個のクラスタのそれぞれに一定の重み u_{li} ただし i = 1, 2, .., N 、 l = 1, 2, .., c をもって所属。チューニングパラメータmが大きいとばらつきが大きくなる
- Rでは
cmeans{e1071}(SVMなどさまざまな機会学習系モデルのパッケージ、データは観測値のみで与える。距離行列は与えられない)fanny{cluster}(クラスタリングの拡張パッケージ、データは観測でも距離行列でも指定できる)FKM{fclust}(ファジィクラスタリング用のパッケージ、データは観測値のみで与える。距離行列は与えられない)
\sum_{l=1}^c \sum_{i=1}^N (u_{li})^m \parallel x_i -\mu_l \parallel^2 \rightarrow min!
ただし
c : 分割数
N : 観測の数
u_{li} : 重み(所属確率)
m : ファジィさを決めるパラメータ
x_i : 観測
\mu_l : クラスタ l の重心
k-harmonic means
- ハードクラスタリング
- 調和平均を使う
- k-meansや混合分布モデルほど初期値に影響されない
- Rではパッケージがない模様
\sum_{i=1}^N \frac{k}{\sum_{l=1}^k \frac{1}{\parallel x_i -\mu_l \parallel^2}} \rightarrow min!
ただし
k : 分割数
N : 観測の数
x_i : 観測
\mu_l : クラスタ l の重心
混合分布モデル/潜在クラスモデル
- 混合分布モデル:特徴量が連続量
- 潜在クラスモデル:特徴量が名義尺度
付けたい分類があらかじめ決まっている場合寄りの手法になる(従属変数あり、教師あり学習)
EMアルゴリズムで推定するので、EMアルゴリズムという手法名にされていることもある。
Rの{mclust}は正規分布の混合分布を前提、{flexmix}は正規分布以外も可能
http://d.hatena.ne.jp/EulerDijkstra/20130618/1371552902
http://nhkuma.blogspot.jp/2013/08/blog-post_13.html
mclustのほうが限定されているが精度は高い?
sparse dataに対するクラスリング
skmeans{skmeans}を使う
変数をクラスタリングする
これまでは観測(レコード、observation)のクラスタリングだったが、
observationではなく変数をクラスタリングするなら{ClustOfVar}パッケージが便利。
質的変数を含むデータでもクラスタリング可能で、非階層型クラスタリング、階層型クラスタリングいずれもできる。
階層型クラスタリング
hclustvar(X.quanti = data[,c(1,6:20)], X.quali = data[,2:5])
非階層型クラスタリング
kmeansvar(X.quanti = data[,c(1,6:20)], init = 3)
※hclustvar()の中のPCAmixdata::recod()にはrename.levelの指定があるので自動的に質的変数のダミー変数に変数名をつけてくれるが、kmeansvar()ではなぜかその指定がなく、質的変数を含むとエラーが発生する(バグ)。
階層型クラスタリングからの非階層型クラスタリング
tree <- hclustvar(X.quanti = data[,c(1,6:20)], init = 3)
plot(tree)
part_init <- cutreevar(tree, 5)$cluster
part <- kmeansvar(X.quanti = data[,c(1,6:20)], init=part_init, matsim=TRUE)
summary(part)
part$sim
データの加工や分析で使うRの使い方 の記事一覧